基因組學:網關設定與執行
 建議變更
建議變更


根據國家人類基因組研究所( "NHGRI"《基因組學》(Genomics)、《基因組學》(Genomics)是一個人所有基因(基因組)的研究、包括這些基因彼此互動、以及與個人環境的互動。」
根據 "NHGRI"「脫氧合核DNA(DNA)是一種化學複合物、內含開發和引導幾乎所有生物體活動所需的指示。DNA分子是由兩股扭曲的配對股組成、通常稱為雙螺旋。」 「生物的完整DNA組稱為基因組。」
排序是決定DNA中基礎的確切順序的程序。當今最常見的排序類型之一、稱為「合成排序」。此技術使用螢光訊號的放射來訂購底座。研究人員可以使用DNA定序來搜尋基因變異、以及在病患仍處於初期階段時、可能在疾病的發展或發展過程中扮演重要角色的任何變異。
從樣本到變體識別、註釋和預測
高層級的基因組學可分為下列步驟。這份清單並非詳盡無遺:
下圖顯示從取樣到變體識別、註釋和預測的程序。
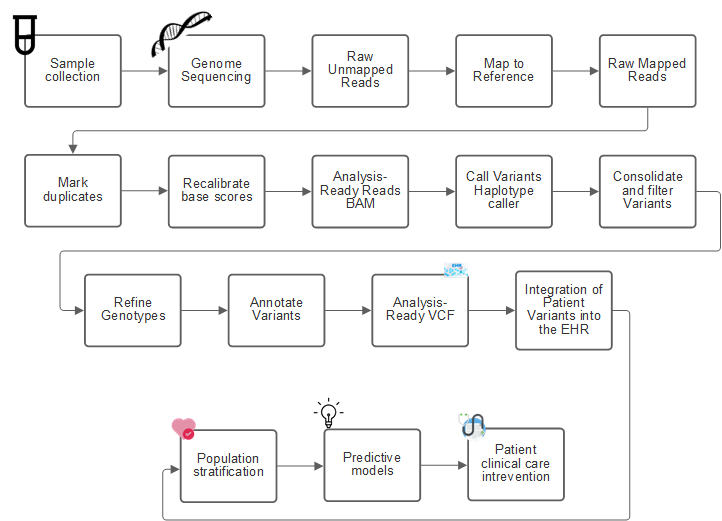
人類基因組項目於2003年4月完成、該項目對公共領域的人類基因組序列進行了非常高質量的模擬。這種參考基因組在基因組能力的研究與開發方面、開始爆炸性發展。幾乎每個人的基因都有其特徵。直到最近、醫師仍利用基因來預測和判斷生兒缺陷、例如單一基因改變所造成的特定繼承模式、例如:粗細胞性貧血。人類基因組專案所提供的寶貴資料、導致基因組功能的現況出現。
基因組學具有廣泛的優點。以下是醫療與生命科學領域的一小組優點:
-
在照護點進行更好的診斷
-
更好的預測
-
精密醫學
-
個人化的治療計畫
-
更佳的疾病監控
-
減少不良事件
-
改善治療方法
-
改善疾病監控
-
有效參與臨床試驗、並根據基因分型、更好地選擇病患進行臨床試驗。
基因組學是 "四頭獸、" 因為資料集生命週期內的運算需求:擷取、儲存、發佈及分析。
基因組分析工具套件(網關)
GAT是以資料科學平台的形式開發 "廣泛研究所"。GATE是一組開放原始碼工具、可進行基因組分析、特別是變種探索、識別、註釋和基因分型。網格連結的好處之一是工具和或命令集可以鏈結在一起、形成完整的工作流程。廣泛機構所面臨的主要挑戰如下:
-
瞭解疾病的根本原因和生物機制。
-
辨識出在疾病根本原因上採取行動的治療介入措施。
-
瞭解從變異到人類生理學的觀察線。
-
建立標準與原則 "架構" 適用於基因組資料呈現、儲存、分析、安全等。
-
標準化並推廣可互通的基因組集合體資料庫(gnomAD)。
-
以基因組為基礎的病患監控、診斷及治療、更精確。
-
協助實作可在症狀出現之前及早預測疾病的工具。
-
建立跨專業合作夥伴社群並賦予其能力、以協助處理生物醫學中最棘手且最重要的問題。
根據GATE和廣義研究所的資料、基因組排序應視為在實驗室中的一種傳輸協定;每項工作都有完整的記錄、最佳化、可重複執行、而且在各個樣本和實驗之間都一致。以下是廣泛研究所建議的一組步驟、如需詳細資訊、請參閱 "網站"。
系統設定FlexPod
基因組學工作負載驗證包括FlexPod 一套完整的功能、可從零開始設定一套完整的基礎架構平台。此平台具備高可用度、可獨立擴充、例如網路、儲存設備和運算都可獨立擴充。FlexPod我們使用下列Cisco驗證設計指南做為參考架構文件、來設定FlexPod 這個環境: "VMware vSphere 7.0與NetApp VMware vCenter 9.7的資料中心FlexPod ONTAP"。請參閱FlexPod 下列的《不完整平台設定摘要:
若要執行FlexPod 程式不實的設定、請完成下列步驟:
-
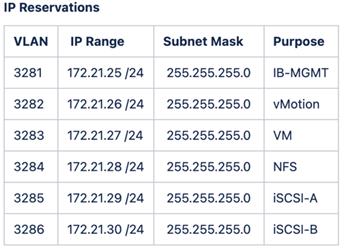
支援下列IP4保留與VLAN、以進行內部測試設定與驗證。FlexPod
-
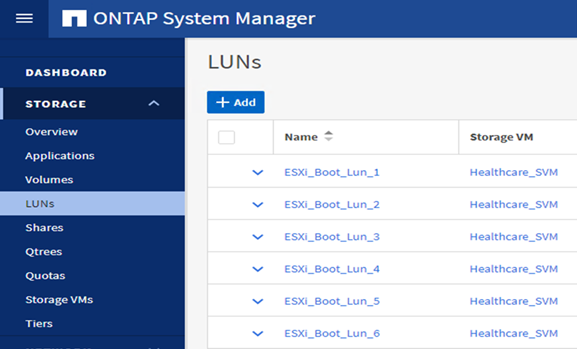
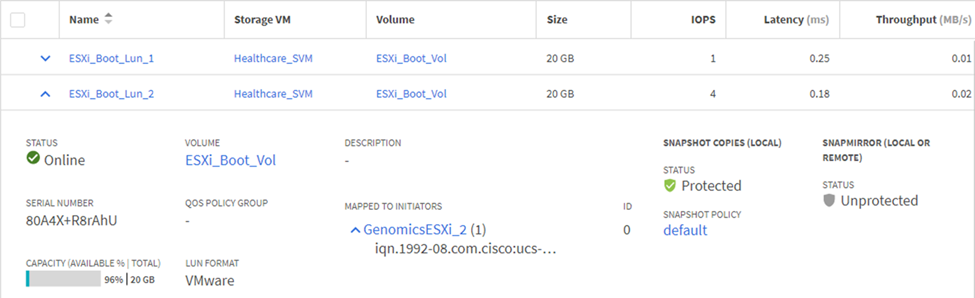
在ONTAP SVM上設定iSCSI型開機LUN。
-
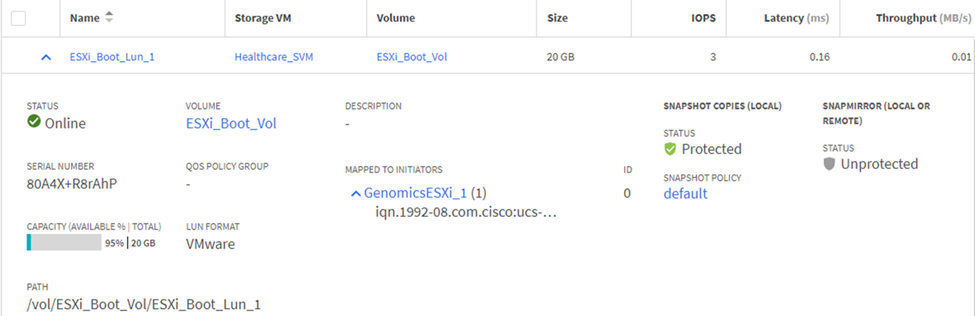
將LUN對應至iSCSI啟動器群組。
-
安裝vSphere 7.0搭配iSCSI開機。
-
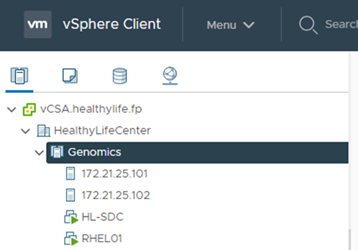
向vCenter登錄ESXi主機。
-
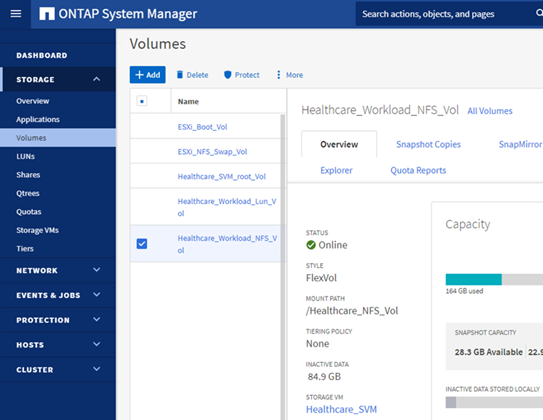
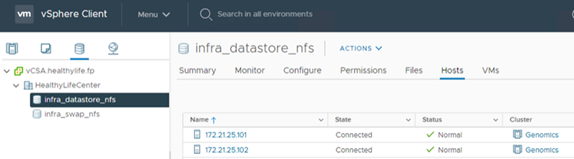
將NFS資料存放區「infra_datastore_nfs」配置到ONTAP 整個儲存區。
-
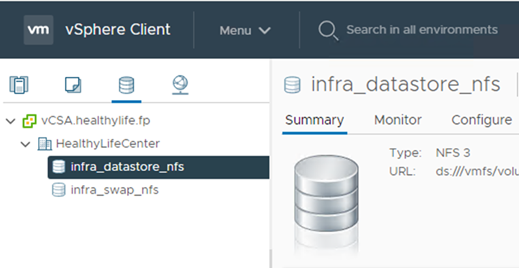
將資料存放區新增至vCenter。
-
使用vCenter將NFS資料存放區新增至ESXi主機。
-
使用vCenter建立Red Hat Enterprise Linux(RHEL)8.3 VM來執行網關。
-
NFS資料存放區會顯示給VM、並掛載於「/mnt/genomics」、用於儲存網關執行檔、指令碼、二進位對齊對應(BAM)檔案、參考檔案、索引檔案、字典檔案及輸出檔案、以供進行變式呼叫。

網關設定與執行
在RedHat Enterprise 8.3 Linux VM上安裝下列先決條件:
-
Java 8或SDK 1.8或更新版本
-
請從廣泛研究所下載網關4.2.0.0 "GitHub網站"。基因組序列資料通常以一系列以索引標籤分隔的Ascii欄的形式儲存。不過、要儲存的空間太多。因此、新的標準演變為BAM(*。bam)檔案。BAM檔案會以壓縮、索引及二進位格式來儲存順序資料。我們 "已下載" 一組公開提供的BAM檔案、可從執行網頁網頁 "公有網域"。我們也下載了索引檔案(\*。Bai)、字典檔案(*)。dict)和參考資料檔案(*。fasta)。
下載後、網關工具套件會有一個Jar檔案和一組支援指令碼。
-
「gatk-package-4.2.0.0-local.jar」執行檔
-
"gatk"指令碼檔案。
我們下載了BAM檔案、以及父、母和子*。bam檔案所組成之系列的對應索引、字典和參考基因組檔案。
Cromwell引擎
Cromwell是開放原始碼引擎、專為科學工作流程所設計、可實現工作流程管理。克倫威爾引擎可在兩個引擎中執行 "模式"、伺服器模式或單一工作流程執行模式。您可以使用來控制Cromwell引擎的行為 "Cromwell引擎組態檔"。
我們使用Cromwell引擎大規模執行工作流程和管線。Cromwell引擎使用方便使用的產品 "工作流程說明語言" (WDL)型指令碼語言。Cromwell也支援第二個工作流程指令碼標準、稱為通用工作流程語言(CML)。在本技術報告中、我們使用WDL。WDL最初由廣義基因組分析管線研究所開發、使用WDL工作流程可以使用多種策略來實作、包括:
-
*線性鏈結*顧名思義、工作#1的輸出會以輸入方式傳送至工作#2。
-
*多重輸入/輸出。*這類似於線性鏈結、因為每個工作都能將多個輸出作為後續工作的輸入傳送。
-
* Scater-GAI.*這是市面上最強大的企業應用程式整合(AI)策略之一、尤其是在事件導向架構中使用時。每項工作都會以分離的方式執行、每項工作的輸出都會整合到最終輸出中。
使用WDL以獨立模式執行網關時、有三個步驟:
-
使用「womitool.jar」驗證語法。
[root@genomics1 ~]# java -jar womtool.jar validate ghplo.wdl
-
產生輸入JSON.
[root@genomics1 ~]# java -jar womtool.jar inputs ghplo.wdl > ghplo.json
-
使用Cromwell引擎和「Cromwell .jar」來執行工作流程。
[root@genomics1 ~]# java -jar cromwell.jar run ghplo.wdl –-inputs ghplo.json
網關可以使用多種方法來執行、本文將探討其中三種方法。
使用Jar檔案執行GK
讓我們來看看使用Hplotype變體呼叫者執行的單一變體呼叫管線。
[root@genomics1 ~]# java -Dsamjdk.use_async_io_read_samtools=false \ -Dsamjdk.use_async_io_write_samtools=true \ -Dsamjdk.use_async_io_write_tribble=false \ -Dsamjdk.compression_level=2 \ -jar /mnt/genomics/GATK/gatk-4.2.0.0/gatk-package-4.2.0.0-local.jar \ HaplotypeCaller \ --input /mnt/genomics/GATK/TEST\ DATA/bam/workshop_1906_2-germline_bams_father.bam \ --output workshop_1906_2-germline_bams_father.validation.vcf \ --reference /mnt/genomics/GATK/TEST\ DATA/ref/workshop_1906_2-germline_ref_ref.fasta
在這種執行方法中、我們使用網關本機執行Jar檔案、使用單一Java命令來叫用Jar檔案、然後將數個參數傳遞給命令。
-
此參數表示我們正在叫用「Haplotypefaller」變體呼叫者管道。
-
「- INPUT」指定輸入BAM檔案。
-
-`-output'以變式呼叫格式(*。vcf)指定變體輸出檔案。 ("參考資料")。
-
利用「參考」參數、我們正在傳遞參考基因組。
執行後、可在區段中找到輸出詳細資料 "使用Jar檔案執行網關的輸出。"
使用./gatk指令碼執行網關
可以使用「/gatk」指令碼來執行GatK工具套件。讓我們來檢查下列命令:
[root@genomics1 execution]# ./gatk \ --java-options "-Xmx4G" \ HaplotypeCaller \ -I /mnt/genomics/GATK/TEST\ DATA/bam/workshop_1906_2-germline_bams_father.bam \ -R /mnt/genomics/GATK/TEST\ DATA/ref/workshop_1906_2-germline_ref_ref.fasta \ -O /mnt/genomics/GATK/TEST\ DATA/variants.vcf
我們會將數個參數傳遞給命令。
-
此參數表示我們正在叫用「Haplotypefaller」變體呼叫者管道。
-
「-I」指定輸入BAM檔案。
-
-o'以變式呼叫格式(*。vcf)指定變體輸出檔案。 ("參考資料")。
-
使用「-R」參數、我們正在傳遞參考基因組。
執行後、可在區段中找到輸出詳細資料 "016e203cf9beada735f224ab14d0b3af"
使用Cromwell引擎執行網關
我們使用Cromwell引擎來管理網關執行。讓我們來檢查命令列及其參數。
[root@genomics1 genomics]# java -jar cromwell-65.jar \ run /mnt/genomics/GATK/seq/ghplo.wdl \ --inputs /mnt/genomics/GATK/seq/ghplo.json
在這裏,我們通過傳遞"-jar"參數來調用Java命令,表示我們打算執行一個Jar文件,例如"Cromwell -65.jar"。傳遞的下一個參數(「run」)表示Cromwell引擎以執行模式執行、其他可能的選項則是「Server mode」(伺服器模式)。下一個參數是「*」。wdl是「執行」模式用來執行管線的參數。下一個參數是要執行之工作流程的輸入參數集。
以下是「gplo.wdll(gplo.wdll)檔案內容的外觀:
[root@genomics1 seq]# cat ghplo.wdl
workflow helloHaplotypeCaller {
call haplotypeCaller
}
task haplotypeCaller {
File GATK
File RefFasta
File RefIndex
File RefDict
String sampleName
File inputBAM
File bamIndex
command {
java -jar ${GATK} \
HaplotypeCaller \
-R ${RefFasta} \
-I ${inputBAM} \
-O ${sampleName}.raw.indels.snps.vcf
}
output {
File rawVCF = "${sampleName}.raw.indels.snps.vcf"
}
}
[root@genomics1 seq]#
以下是對應的Json檔案、其中包含對Cromwell引擎的輸入。
[root@genomics1 seq]# cat ghplo.json
{
"helloHaplotypeCaller.haplotypeCaller.GATK": "/mnt/genomics/GATK/gatk-4.2.0.0/gatk-package-4.2.0.0-local.jar",
"helloHaplotypeCaller.haplotypeCaller.RefFasta": "/mnt/genomics/GATK/TEST DATA/ref/workshop_1906_2-germline_ref_ref.fasta",
"helloHaplotypeCaller.haplotypeCaller.RefIndex": "/mnt/genomics/GATK/TEST DATA/ref/workshop_1906_2-germline_ref_ref.fasta.fai",
"helloHaplotypeCaller.haplotypeCaller.RefDict": "/mnt/genomics/GATK/TEST DATA/ref/workshop_1906_2-germline_ref_ref.dict",
"helloHaplotypeCaller.haplotypeCaller.sampleName": "fatherbam",
"helloHaplotypeCaller.haplotypeCaller.inputBAM": "/mnt/genomics/GATK/TEST DATA/bam/workshop_1906_2-germline_bams_father.bam",
"helloHaplotypeCaller.haplotypeCaller.bamIndex": "/mnt/genomics/GATK/TEST DATA/bam/workshop_1906_2-germline_bams_father.bai"
}
[root@genomics1 seq]#
請注意、Cromwell使用記憶體內建資料庫來執行。執行後、可在一節中看到輸出記錄 "使用Cromwell引擎執行網關的輸出。"
如需如何執行網關的完整步驟集、請參閱 "GK文件"。


